Activités de l'équipe
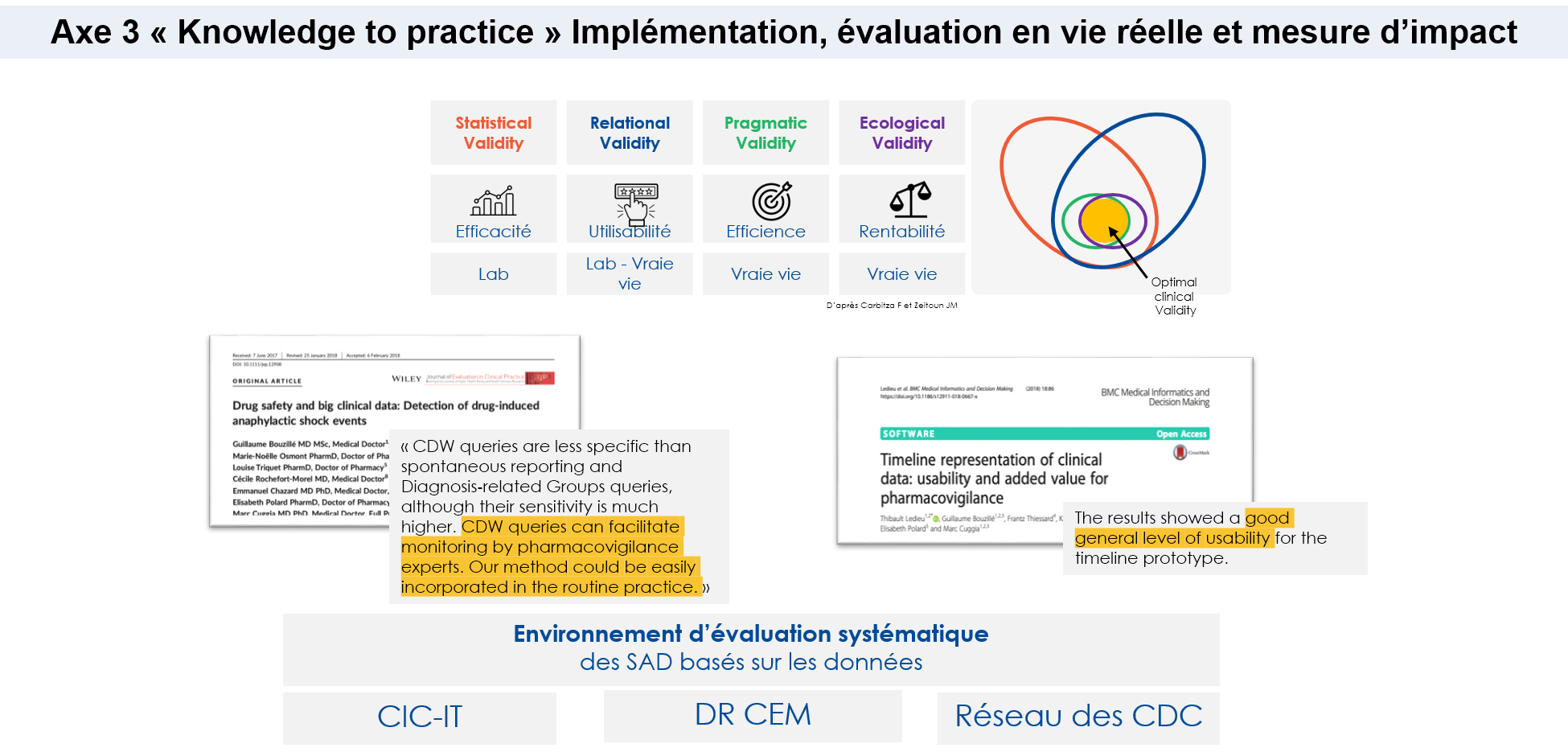 La troisième activité concerne l’implémentation, évaluation en vie réelle et mesure d’impact des connaissances et modèles produits par l’axe 2.
Une fois les modèles disponibles et validés dans un contexte de recherche, il est nécessaire de planifier leur mise en œuvre dans des outils de soutien clinique, qui complètent directement la pratique clinique au chevet du patient. Dans ce contexte, l'équipe DOMASIA est chargée d'aider les partenaires industriels, ainsi que d'incorporer les produits de la recherche dans des outils cliniquement conformes et validés par leur mise en œuvre en situation réelle. L'intégration des systèmes d'aide à la décision demande en plus des mesures de validité statistique, l'évaluation d'autres dimensions, telles que la sécurité, l'efficacité et la satisfaction dans des contextes de vie réelle.
Pour réaliser ces activités, nous bénéficions du lien fort de l'équipe avec le CIC-IT local et de sa participation au réseau CIC-IT, ainsi que de l'implication directe des départements de recherche clinique. Les projets en cours et futurs ont pour but de ou de continuer à produire des outils prêts à l'emploi, qui ont montré des performances prometteuses non seulement dans des contextes expérimentaux, mais aussi dans des contextes de vie réelle.
Finalement, une dernière tâche est nécessaire : la surveillance continue du système afin de pouvoir documenter les changements de performance. Cette performance deviendra une nouvelle donnée pour alimenter le système et fermer la boucle du cycle d'apprentissage. Notre objectif est d'aborder la question de l'amélioration des systèmes, mais aussi et surtout de réinjecter les données de la recherche dans les soins et la vie réelle.
La troisième activité concerne l’implémentation, évaluation en vie réelle et mesure d’impact des connaissances et modèles produits par l’axe 2.
Une fois les modèles disponibles et validés dans un contexte de recherche, il est nécessaire de planifier leur mise en œuvre dans des outils de soutien clinique, qui complètent directement la pratique clinique au chevet du patient. Dans ce contexte, l'équipe DOMASIA est chargée d'aider les partenaires industriels, ainsi que d'incorporer les produits de la recherche dans des outils cliniquement conformes et validés par leur mise en œuvre en situation réelle. L'intégration des systèmes d'aide à la décision demande en plus des mesures de validité statistique, l'évaluation d'autres dimensions, telles que la sécurité, l'efficacité et la satisfaction dans des contextes de vie réelle.
Pour réaliser ces activités, nous bénéficions du lien fort de l'équipe avec le CIC-IT local et de sa participation au réseau CIC-IT, ainsi que de l'implication directe des départements de recherche clinique. Les projets en cours et futurs ont pour but de ou de continuer à produire des outils prêts à l'emploi, qui ont montré des performances prometteuses non seulement dans des contextes expérimentaux, mais aussi dans des contextes de vie réelle.
Finalement, une dernière tâche est nécessaire : la surveillance continue du système afin de pouvoir documenter les changements de performance. Cette performance deviendra une nouvelle donnée pour alimenter le système et fermer la boucle du cycle d'apprentissage. Notre objectif est d'aborder la question de l'amélioration des systèmes, mais aussi et surtout de réinjecter les données de la recherche dans les soins et la vie réelle.
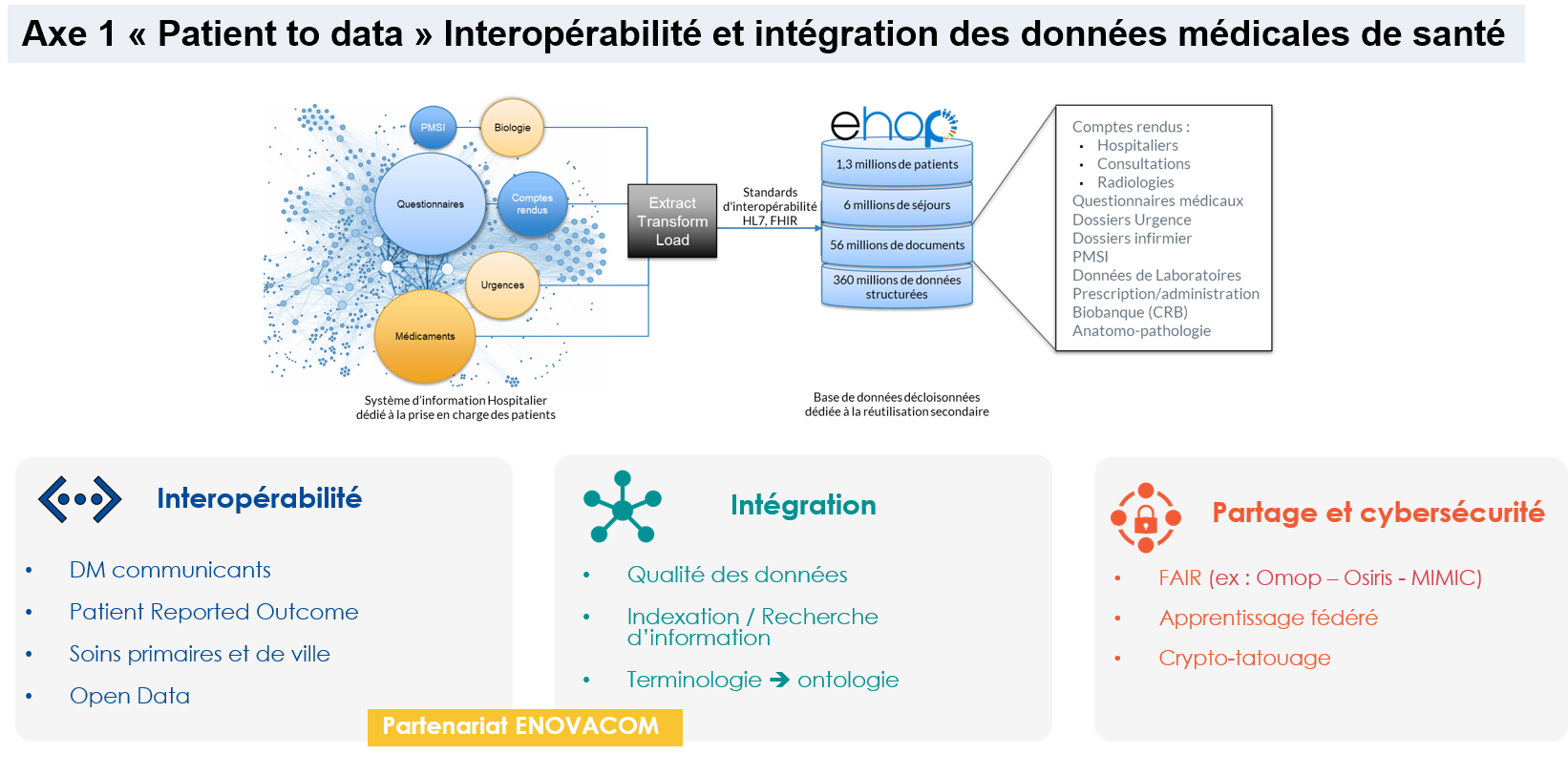
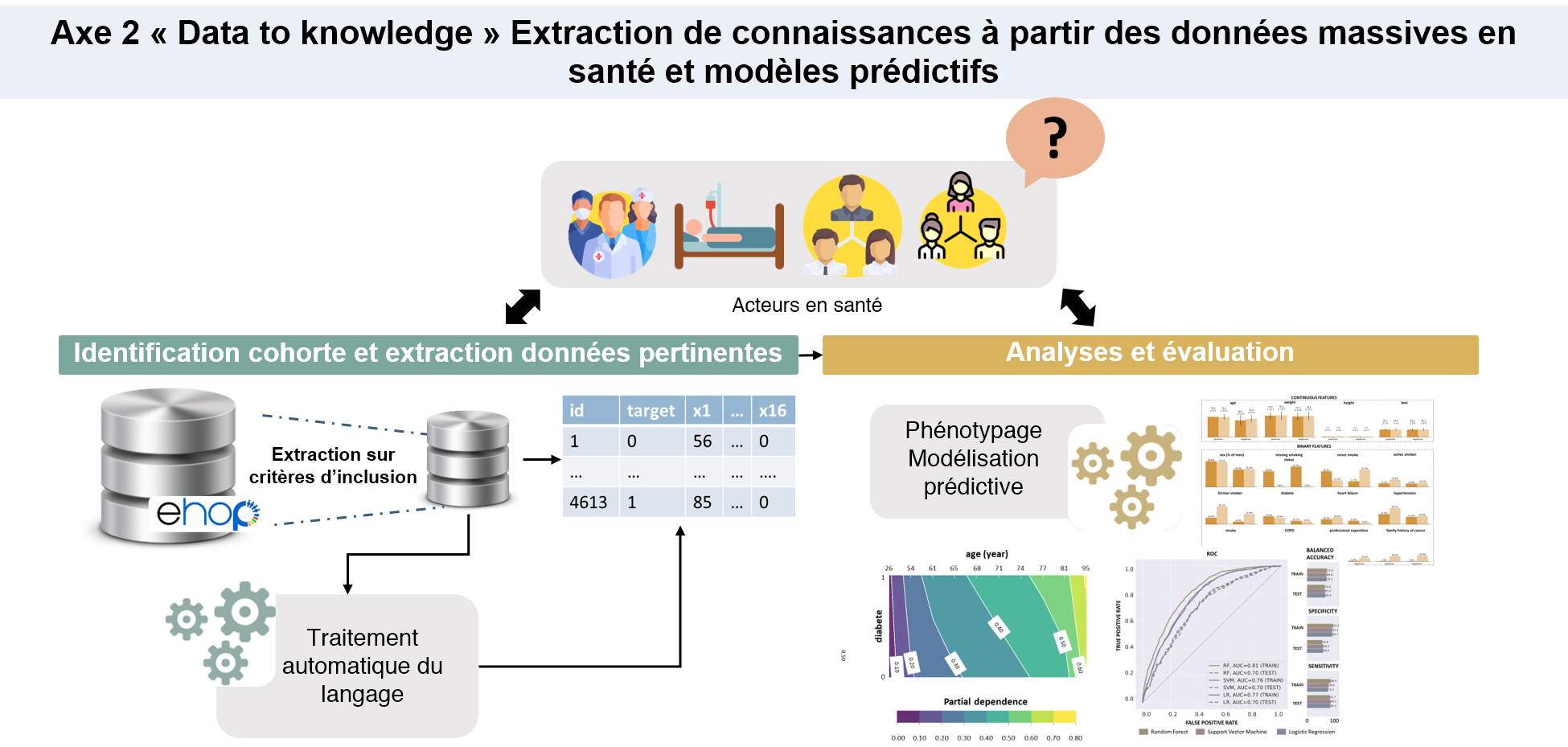
 La troisième activité concerne l’implémentation, évaluation en vie réelle et mesure d’impact des connaissances et modèles produits par l’axe 2.
Une fois les modèles disponibles et validés dans un contexte de recherche, il est nécessaire de planifier leur mise en œuvre dans des outils de soutien clinique, qui complètent directement la pratique clinique au chevet du patient. Dans ce contexte, l'équipe DOMASIA est chargée d'aider les partenaires industriels, ainsi que d'incorporer les produits de la recherche dans des outils cliniquement conformes et validés par leur mise en œuvre en situation réelle. L'intégration des systèmes d'aide à la décision demande en plus des mesures de validité statistique, l'évaluation d'autres dimensions, telles que la sécurité, l'efficacité et la satisfaction dans des contextes de vie réelle.
Pour réaliser ces activités, nous bénéficions du lien fort de l'équipe avec le CIC-IT local et de sa participation au réseau CIC-IT, ainsi que de l'implication directe des départements de recherche clinique. Les projets en cours et futurs ont pour but de ou de continuer à produire des outils prêts à l'emploi, qui ont montré des performances prometteuses non seulement dans des contextes expérimentaux, mais aussi dans des contextes de vie réelle.
Finalement, une dernière tâche est nécessaire : la surveillance continue du système afin de pouvoir documenter les changements de performance. Cette performance deviendra une nouvelle donnée pour alimenter le système et fermer la boucle du cycle d'apprentissage. Notre objectif est d'aborder la question de l'amélioration des systèmes, mais aussi et surtout de réinjecter les données de la recherche dans les soins et la vie réelle.
La troisième activité concerne l’implémentation, évaluation en vie réelle et mesure d’impact des connaissances et modèles produits par l’axe 2.
Une fois les modèles disponibles et validés dans un contexte de recherche, il est nécessaire de planifier leur mise en œuvre dans des outils de soutien clinique, qui complètent directement la pratique clinique au chevet du patient. Dans ce contexte, l'équipe DOMASIA est chargée d'aider les partenaires industriels, ainsi que d'incorporer les produits de la recherche dans des outils cliniquement conformes et validés par leur mise en œuvre en situation réelle. L'intégration des systèmes d'aide à la décision demande en plus des mesures de validité statistique, l'évaluation d'autres dimensions, telles que la sécurité, l'efficacité et la satisfaction dans des contextes de vie réelle.
Pour réaliser ces activités, nous bénéficions du lien fort de l'équipe avec le CIC-IT local et de sa participation au réseau CIC-IT, ainsi que de l'implication directe des départements de recherche clinique. Les projets en cours et futurs ont pour but de ou de continuer à produire des outils prêts à l'emploi, qui ont montré des performances prometteuses non seulement dans des contextes expérimentaux, mais aussi dans des contextes de vie réelle.
Finalement, une dernière tâche est nécessaire : la surveillance continue du système afin de pouvoir documenter les changements de performance. Cette performance deviendra une nouvelle donnée pour alimenter le système et fermer la boucle du cycle d'apprentissage. Notre objectif est d'aborder la question de l'amélioration des systèmes, mais aussi et surtout de réinjecter les données de la recherche dans les soins et la vie réelle.